Программа для просмотра мрт для андроид
Note: This article is an Evergreen article. It will receive continues update about the listed applications. You can save it and return to it as a updated resource.
DICOM (Digital Imaging and Communications in Medicine) is the international standard to transmit, store, retrieve, print, process, and display medical imaging information. The Following is a list of the best free DICOM viewers for doctors.
We have covered several categories for DICOM viewers, here what we covered so far:
- Free & Open source DICOM viewers for Windows
- Free Online Web-based DICOM Viewers & Free DICOM cloud services
- Free & Open source DICOM viewers for Mac OS X
- Open source Browser & Web-based DICOM Viewers Projects
- Best Free & Open source PACS Clients & DICOM Workstations for Windows, Mac OSX, & Linux
- Cross-platform Open source DICOM viewers for Windows, Linux, & Mac OSX
Free Online Web-based DICOM viewers are online cloud services that display upload or remote DICOM files or folders with DICOM specific browser. Here is a compiled list of cloud DICOM viewers.
In this article we have covered the best free and open source DICOM viewers for many platforms. This list is always updated with emerging and new software regularly, so stay tuned.
Free & Open source enterprise-grade DICOM viewers
 Navegatium DICOM Viewer
Navegatium DICOM Viewer
Incredibly easy to use, Navegatium has an absolutely intuitive touchscreen interface, works perfectly on Windows 10 and Windows 8.1 tablets, laptops and PCs and can be installed directly from Windows Store. It makes out list of Best Free & Open source PACS Clients & DICOM Workstations, because of its outstanding features.
It is capable of 2D, 3D and X-Ray reconstructions with DICOM images.
We have written a snap preview about Nevegatium, you can read it here.
- Platform: Windows 10 and Windows 8.1
- Navegatium DICOM Viewer website
2- Sante DICOM Viewer Free version
 Sante DICOM Viewer Free
Sante DICOM Viewer Free
Sante DICOM Viewer Free can be used as a stand-alone desktop application, but also it is suitable for DICOM CD/DVD distribution. It runs from CD/DVD without installation, it does not have any additional software requirements like .NET or Java run-time libraries, and opens automatically the DICOMDIR file in the root of the CD/DVD.
 Sante DICOM Viewer Free
Sante DICOM Viewer Free
Sante DICOM Viewer Free supports all the modalities (CT, MR, US, CR, NM, XA, MG, DX etc.), all the manufacturers, and all the DICOM file types (DICOM 3.0/NEMA 2).
- Platform: Windows
- Sante DICOM Viewer free
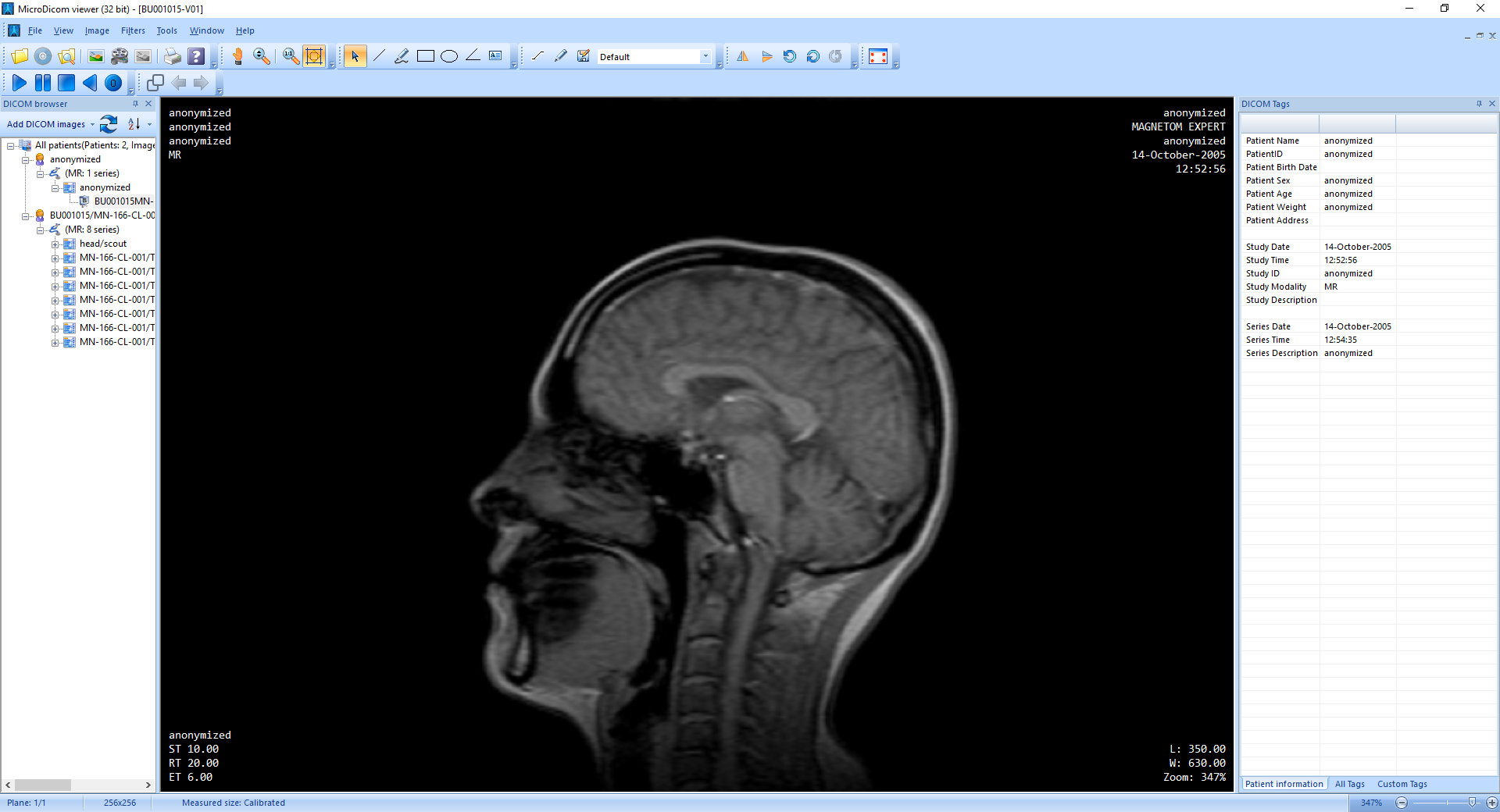 MicroDICOM
MicroDICOM
MicroDicom is an application for primary processing and preservation of medical images in DICOM format.
It is equipped with most common tools for manipulation of DICOM images and it has an intuitive user interface. It is free for non-commercial use.
MicroDicom has been features in our list: Best Free & Open source PACS Clients & DICOM Workstations
- Platform: Windows
- MicroDICOM website
- MicroDicom — free DICOM viewer software for Windows hits 1 million downloads.
4- Horos DICOM Project: Open source DICOM viewer for Mac OSX
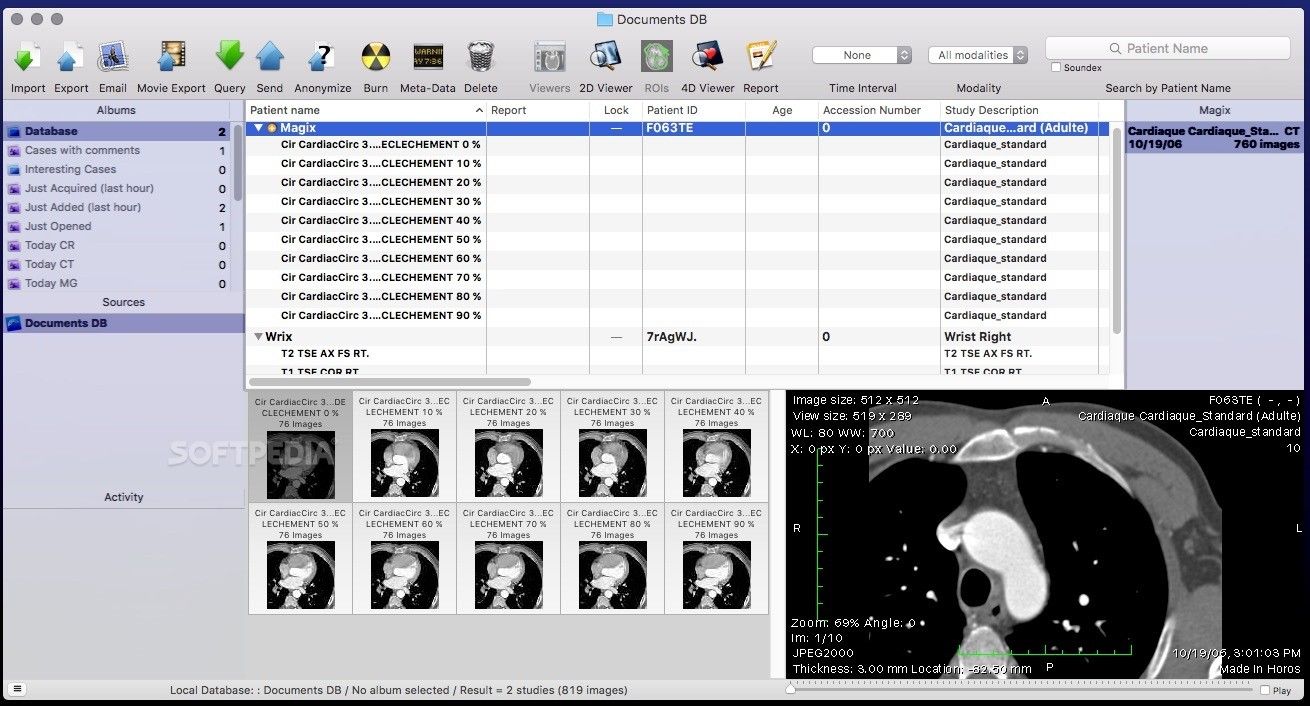 Horos DICOM
Horos DICOM
Horos is a free, open source medical image viewer. The goal of the Horos Project is to develop a fully functional, 64-bit medical image viewer for OS X. Horos is based upon OsiriX and other open source medical imaging libraries. Horos is made available under the GNU Lesser General Public License, Version 3 (LGPL-3.0).
Horos has been featured several times in our list reviews:
- Open source DICOM Viewers
- Open source Free DICOM Viewers for Mac OSX
- Best Free & Open source PACS Clients & DICOM Workstations
- Platform: Mac OSX
- Horos DICOM viewer website, download
- Horos DICOM viewer Medevel’s review
5- JiveX DICOM Viewer
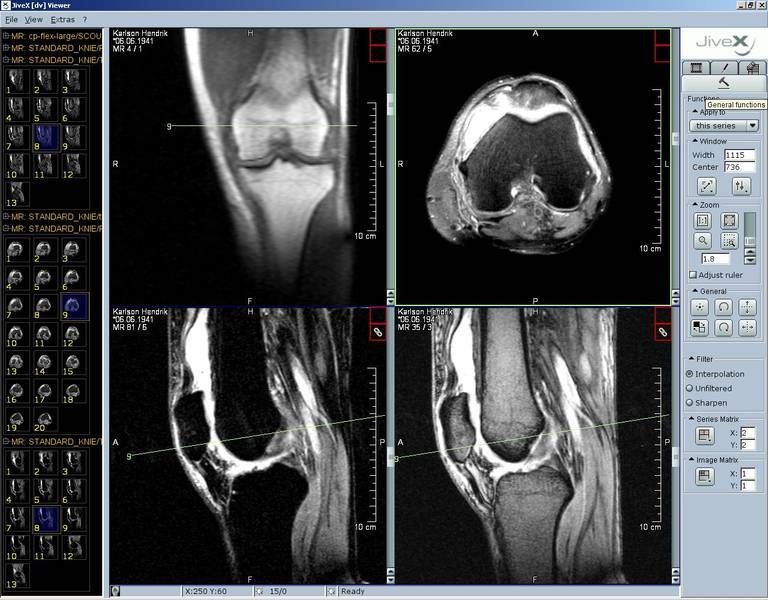 JiveX DICOM Viewer — src
JiveX DICOM Viewer — src
This free DICOM Viewer offers not only to review radiology images but also other types of medical data such as ECGs or information in pdf files – as long as the data is available in DICOM format. Thus the DICOM Viewer provides all users with access to important information far beyond radiology.
Data can be loaded onto the Viewer from an internal folder or from external media such as CD, DVD or stick. Unlike the licensed Review Client the free DICOM Viewer cannot be linked to the PACS. Technical service is not included.
It is available for non-commercial use (research or educational purposes).
- Platform: Windows
- JiveX DICOM Viewer Review
- JiveX DICOM Viewer website
AMIDE is a completely free and open source tool for viewing, analyzing, and registering volumetric medical imaging data sets.
 AMIN DICOM src
AMIN DICOM src
- Platform: Linux, Mac OSX, Windows
- AMIDE DICOM viewer website
7- 3DimViewer
3DimViewer is a lightweight free and opensource 3D viewer of medical DICOM datasets that is distributed as open source software.
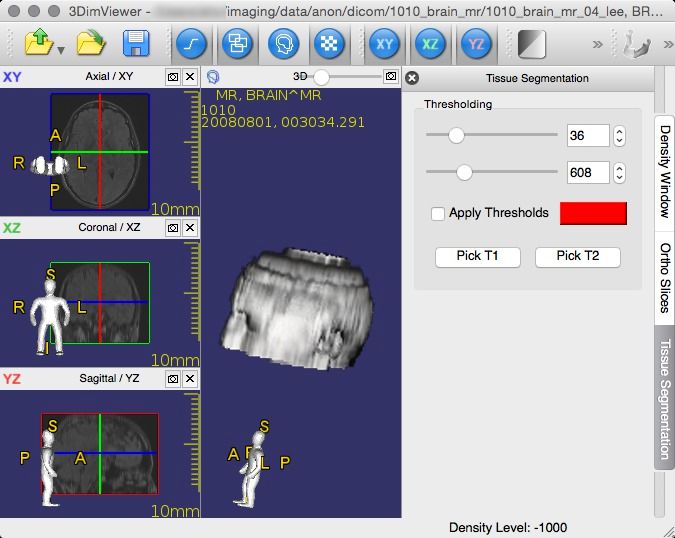 3DimViewer — src
3DimViewer — src
- Platform: Linux, Mac OSX , Windows
- 3DimViewer Link
8- postDICOM
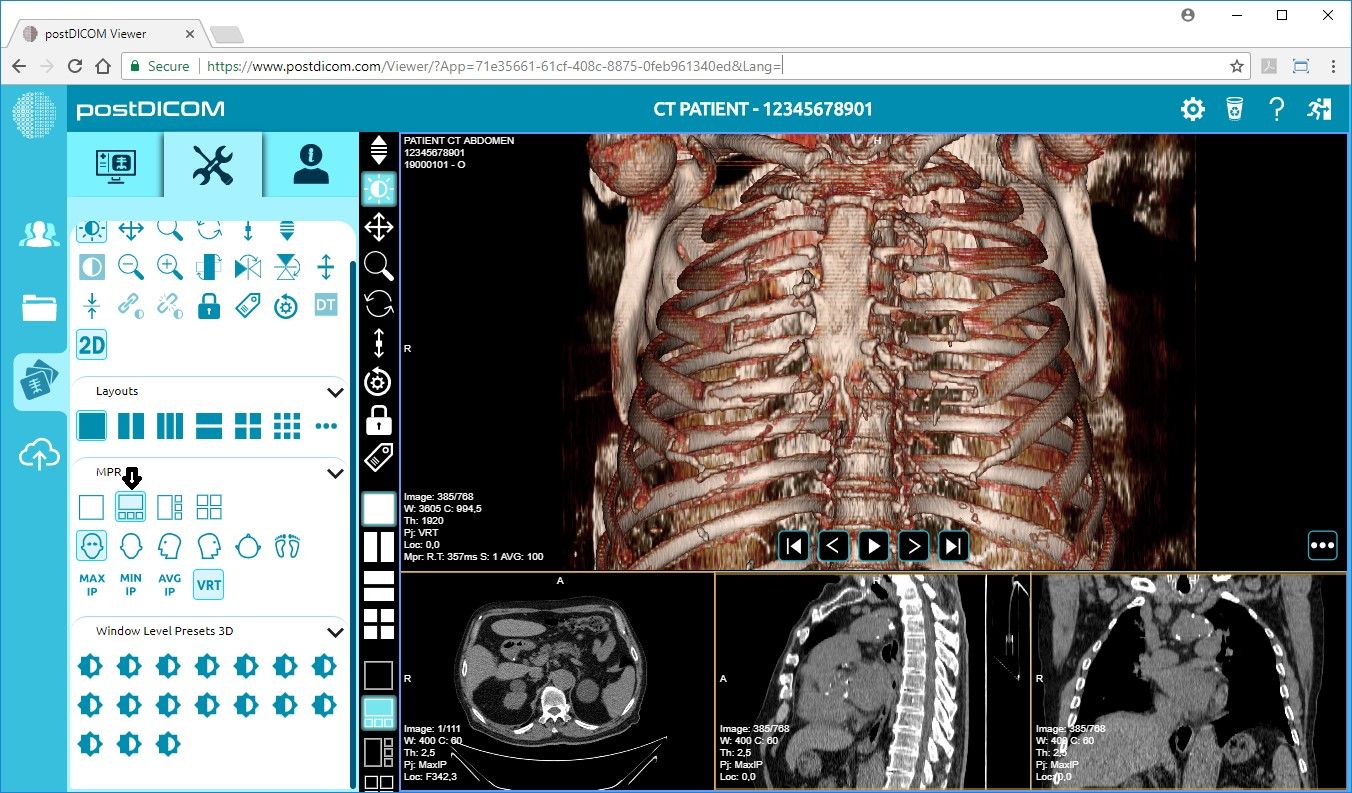 postDICOM: Online DICOM Viewer
postDICOM: Online DICOM Viewer
postDICOM is a secure environment for patients, doctors, clinics and hospitals. Using postDICOM, users can Post medical DICOM images (computed tomography, magnetic resonance imaging, ultrasound, digital radiography etc.) and clinical documents (scanned documents, reports, patient’s history etc.) to cloud. Later, users can view and share these documents with doctors and medical groups. Uploaded DICOM files are streamed using sophisticated HTML5 zero footprint lossless DICOM viewer.
- Platform: Cloud-based
- postDICOM
9- SMILI DICOM viewer
The Simple Medical Imaging Library Interface (SMILI), is an open-source, light-weight and easy-to-use medical imaging viewer and library for all major operating systems. The main sMILX application features for viewing n-D images, vector images, DICOMs, anonymizing, shape analysis and models/surfaces with easy drag and drop functions. It also features a number of standard processing algorithms for smoothing, thresholding, masking etc. images and models, both with graphical user interfaces and/or via the command-line. SMILI is in active development by his developers.
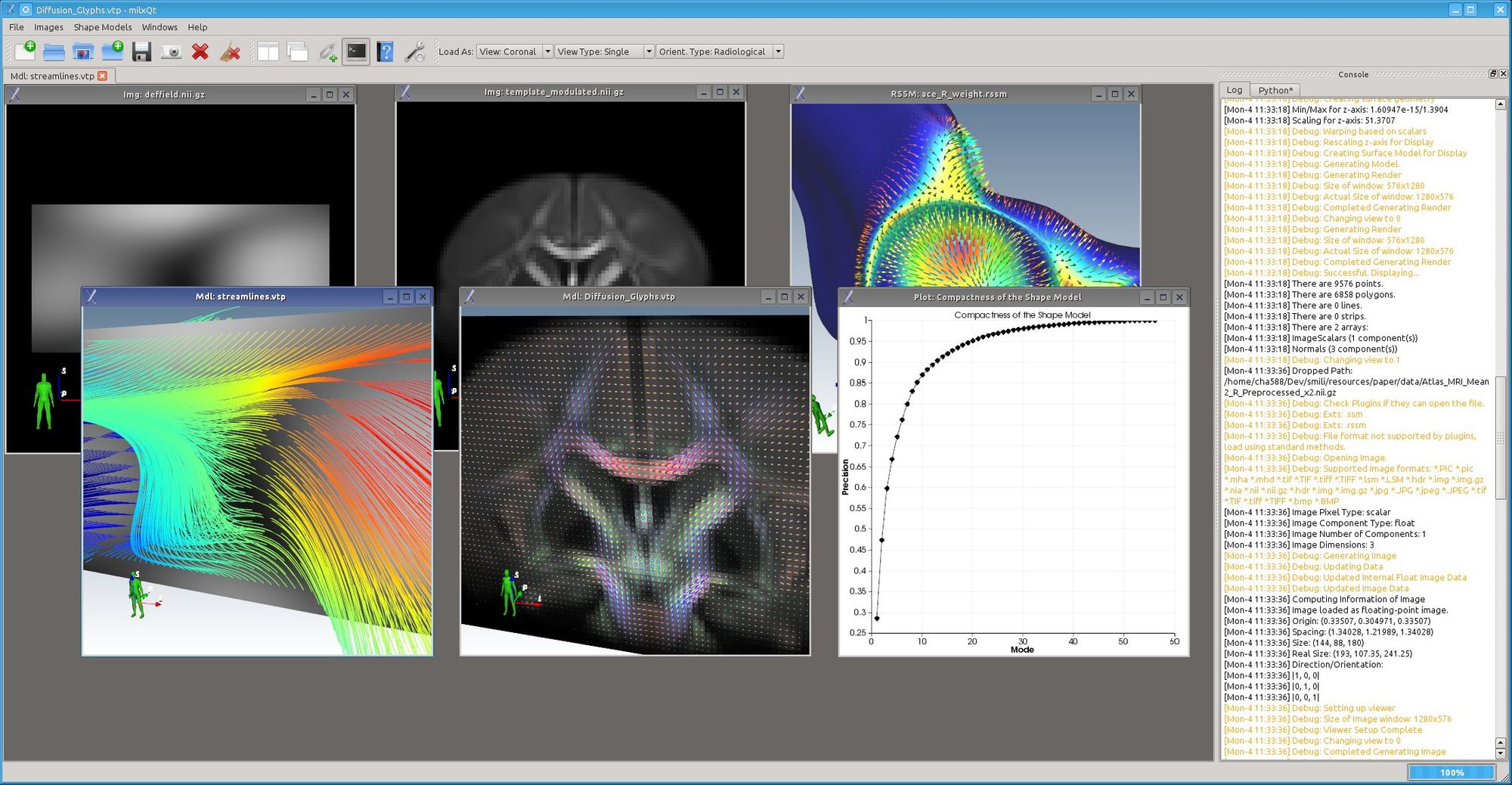 SMILI DICOM viewer
SMILI DICOM viewer
- Platform: Linux, Mac OSX, Windows
- Website, Download
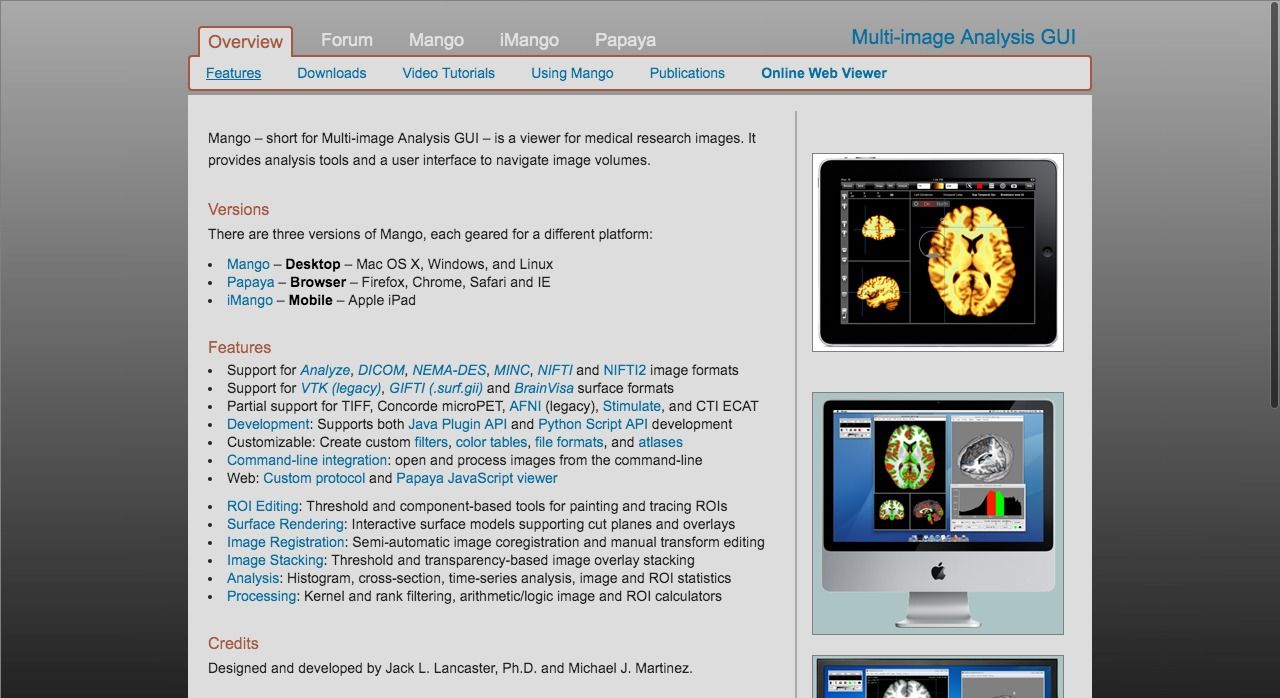 Mongo website
Mongo website
Mango – short for Multi-image Analysis GUI – is a viewer for medical research images. It provides analysis tools and a user interface to navigate image volumes.
Mango is available for use, as is, free of charge, for educational and scientific, non-commercial purposes. Mongo also made it in our featured list as one of the : Best Free & Open source PACS Clients & DICOM Workstations for Windows, Mac OSX, & Linux.
- Platform: Linux, Mac, Windows
- Link: https://ric.uthscsa.edu/mango/
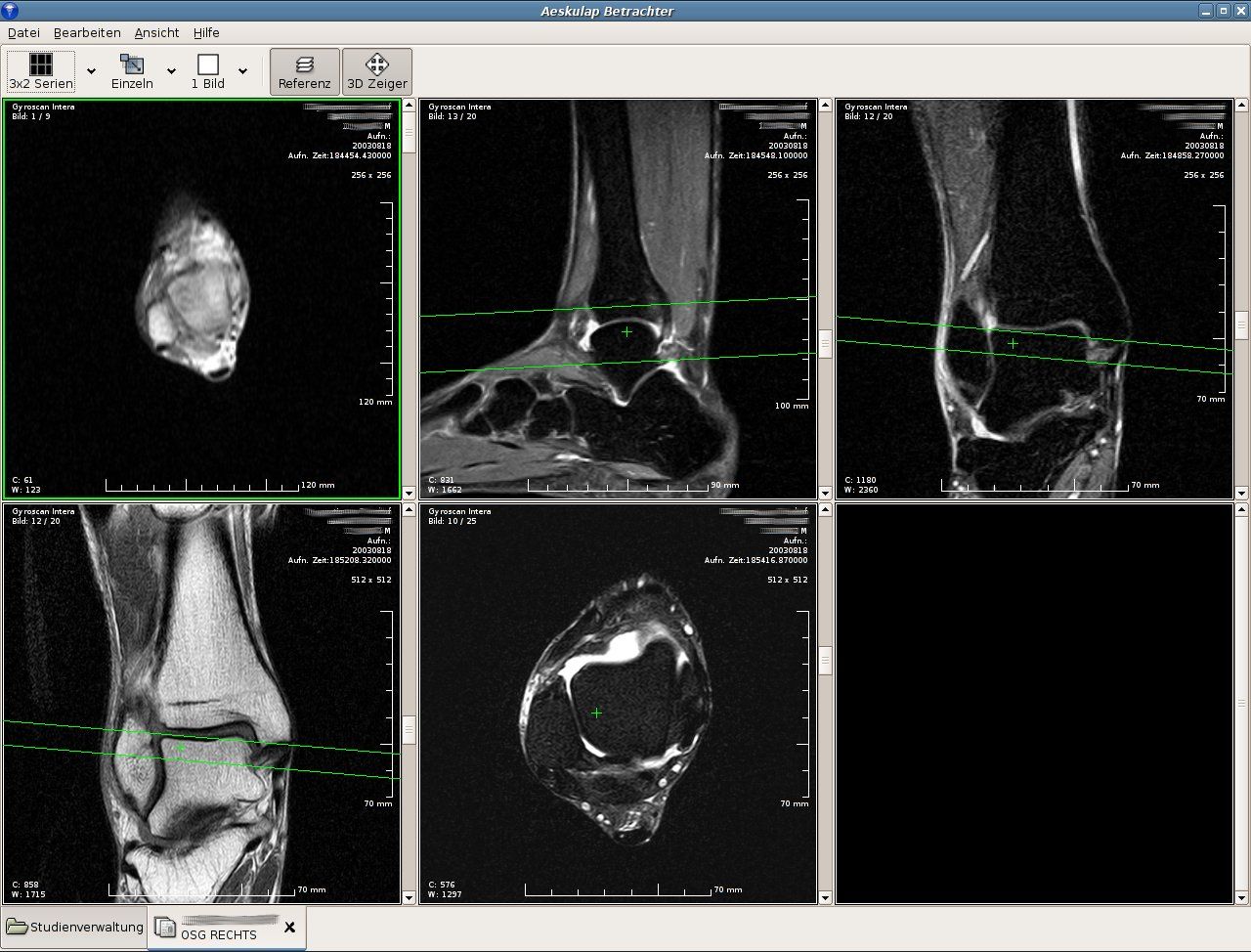
Aeskulap is a very famous DICOM mmanager/ and viewer on Ubuntu/ Debian linux.
Platform : Linux
https://aeskulap.nongnu.org/
12 — NextCloud : Cloud based DICOM Viewer
 NextCloud : Cloud based DICOM Viewer
NextCloud : Cloud based DICOM Viewer
NextCloud is a free opensource self-hosted cloud file sharing solution, It provides security, solid user management and set of desktop and mobile clients which support synchronisation. NextCloud AppStore has DICOM viewer which is full featured DICOM viewer ready to work for teams and hospitals.
Review : NextCloud : Free & open source self-hosted cloud files sharing and healthcare ready platform
Platforms
- Server : Linux, Clients : Mac OSX, Windows, Linux. Mobile Apps Android and iPhone, web browsers desktop and mobile.
13 — Athena DICOM Viewer
Free DICOM viewer
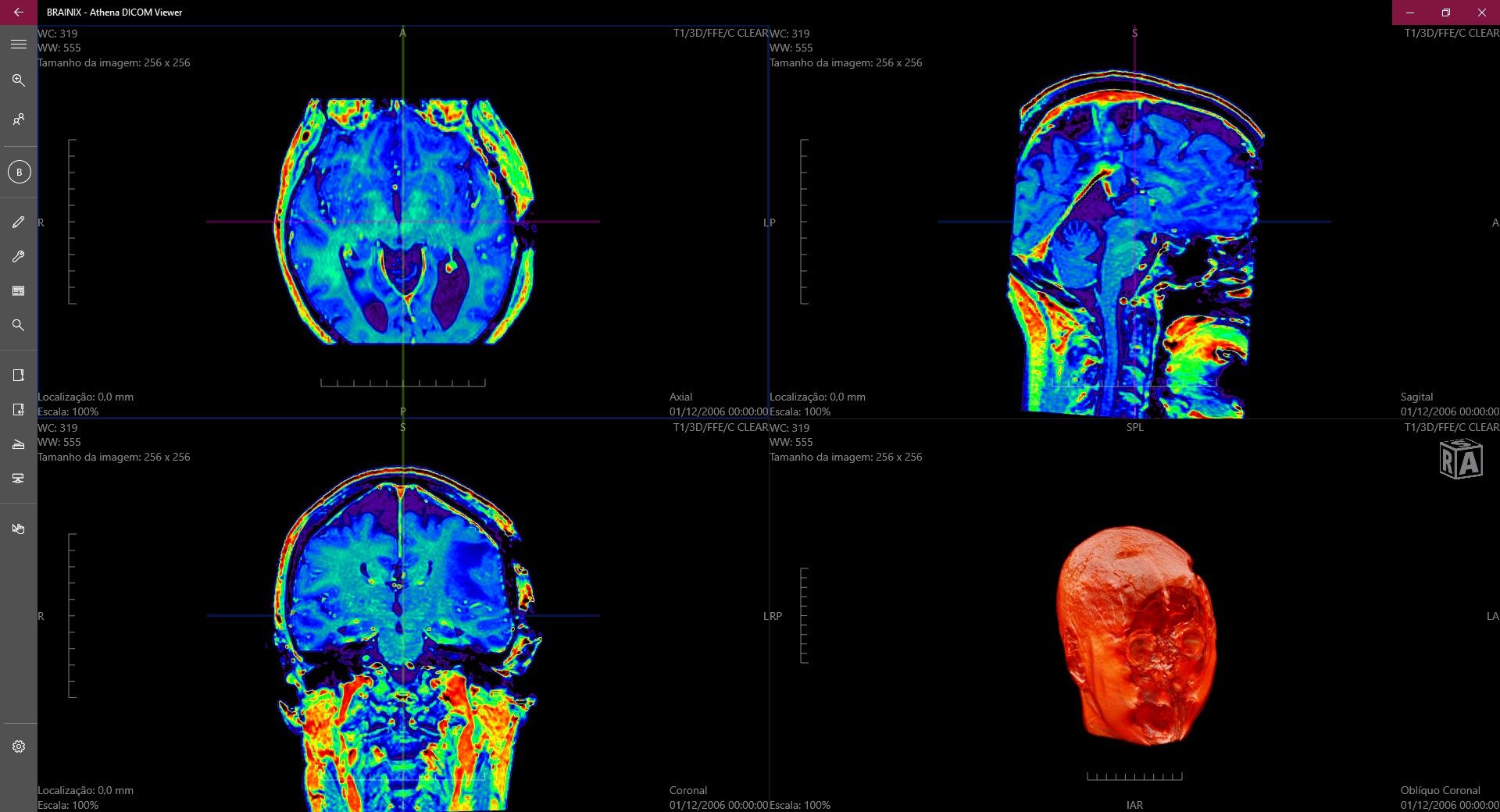
Athena is a lightweight DICOM software, designed to display DICOM images on Windows machines. It’s available in the Windows app store and offers an in-app purchase for additional options an features. Athena has a very simple custom design built for touch screens including Windows Tablets and wide resolution touch screens. Athena Lite edition comes with one month free for Athena pro editions which can be extended later with purchase. Athena was built by native windows technology so it’s expected to be memory wise with smooth performance.
- Platform : Windows
- Athena DICOM viewer download
14 — ClearCanvas DICOM server and DICOM Viewer
ClearCanvas is an open source medical imaging project featuring DICOM server and features-rich DICOM viewer for Windows with DICOM viewer.
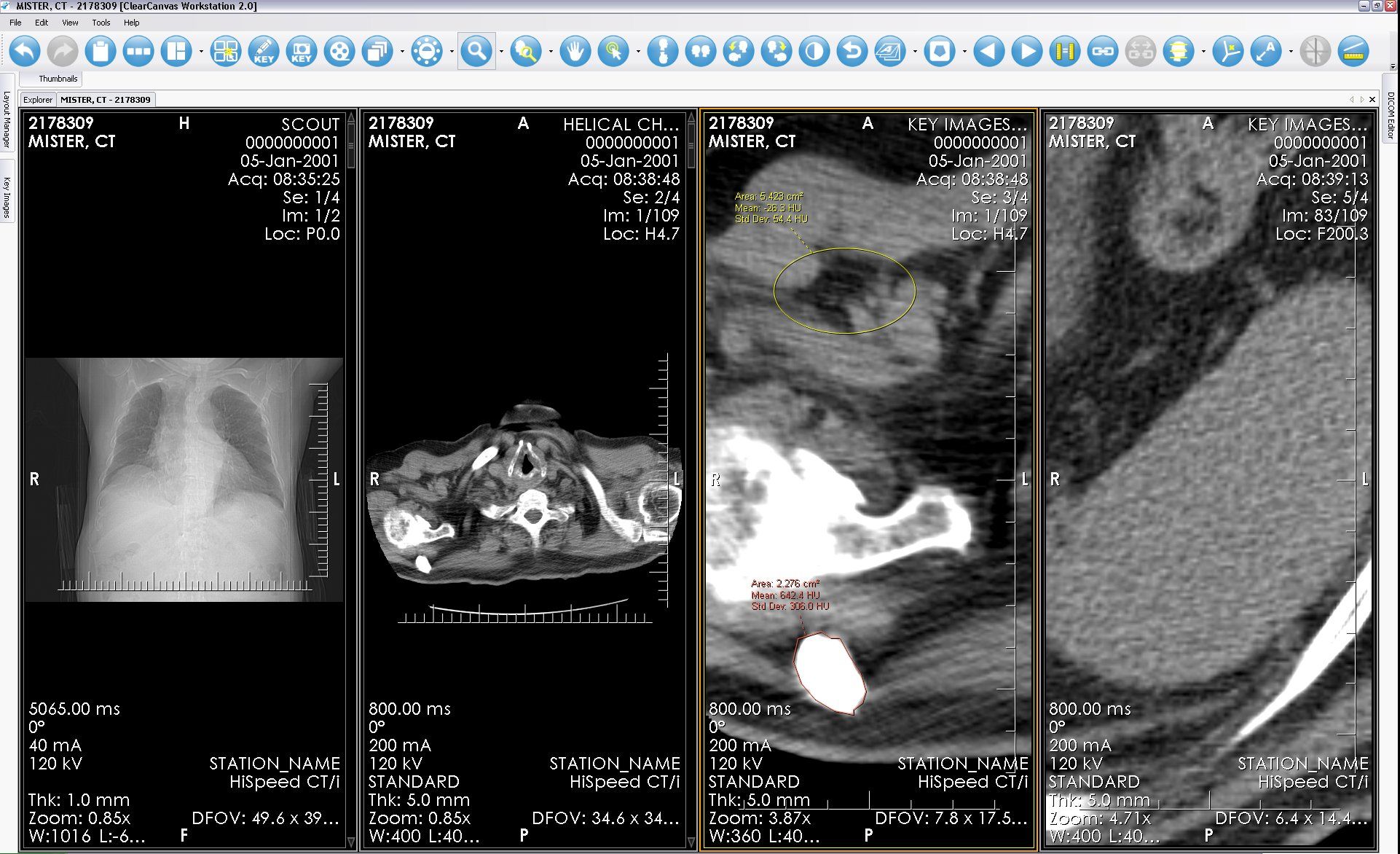 ClearCanvas DICOM
ClearCanvas DICOM
- Platform : Windows , Web viewer
- ClearCanvas Website , ClearCanvas Sourceforge
15- MatrixUser
MatrixUser is a lightweight GUI-based program for multi-dimensional data processing and analysis for medical imaging.
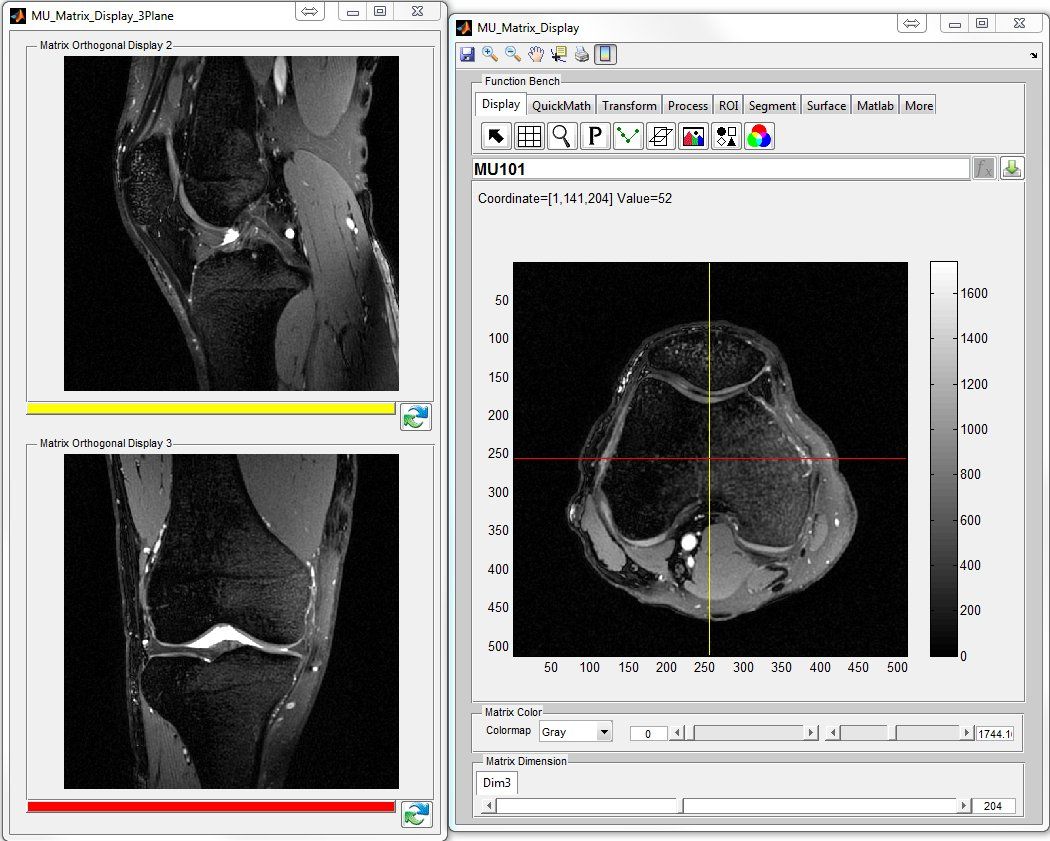
- Highly Interactive Graphical User Interface
- Multi-dimensional Matrix Display and Analysis in Matlab
- Multiple Data Type Conversion for DICOM, DICOM (Batch), NIFTI, MAT and Binary
- Nice DICOM Viewer and Convertor
- ROI Analysis, Segmentation, 3D Slicer and More Functions
MatrixUser website, SourceForge
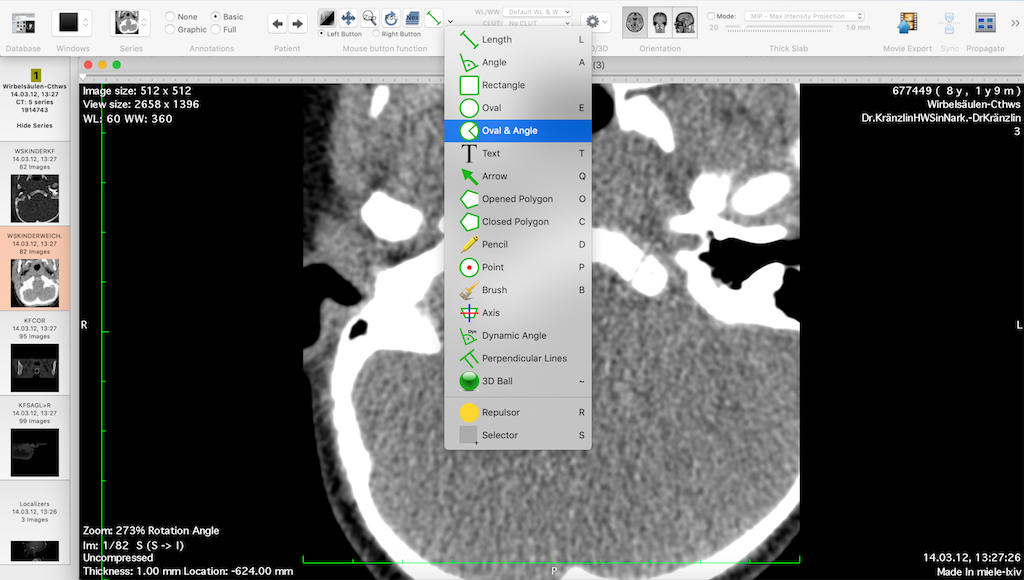
Miele-LXIV is an open source DICOM workstation and viewer for Mac OSX, It’s completely native solution built by apple technologies, It supports 10.9 macOSX version and later, which means it’ll work on old Mac OSX machines.
Miele-LXIV has many features that we have covered in this review.
17- SIVIC: Open source DICOM MR Spectroscopy Framework for Researchers, and Radiologists
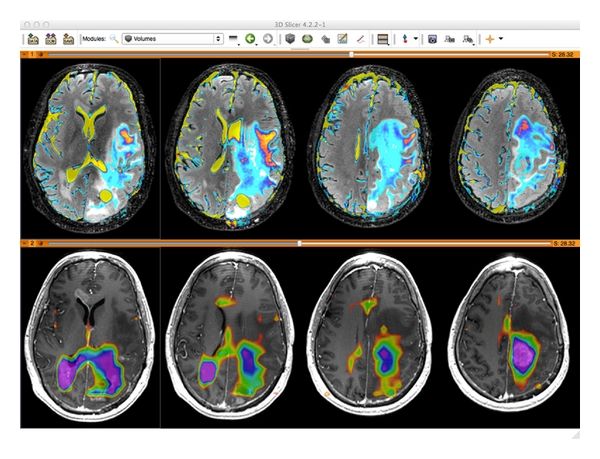 SIVIC: Open source DICOM MR Spectroscopy Framework for Researchers, and Radiologists
SIVIC: Open source DICOM MR Spectroscopy Framework for Researchers, and Radiologists
SIVIC is a customised DICOM MR Spectroscopy framework for researchers and radiologists, created by Nelson Lab.
18- Gimias: Medical Imaging Framework/ Simulation
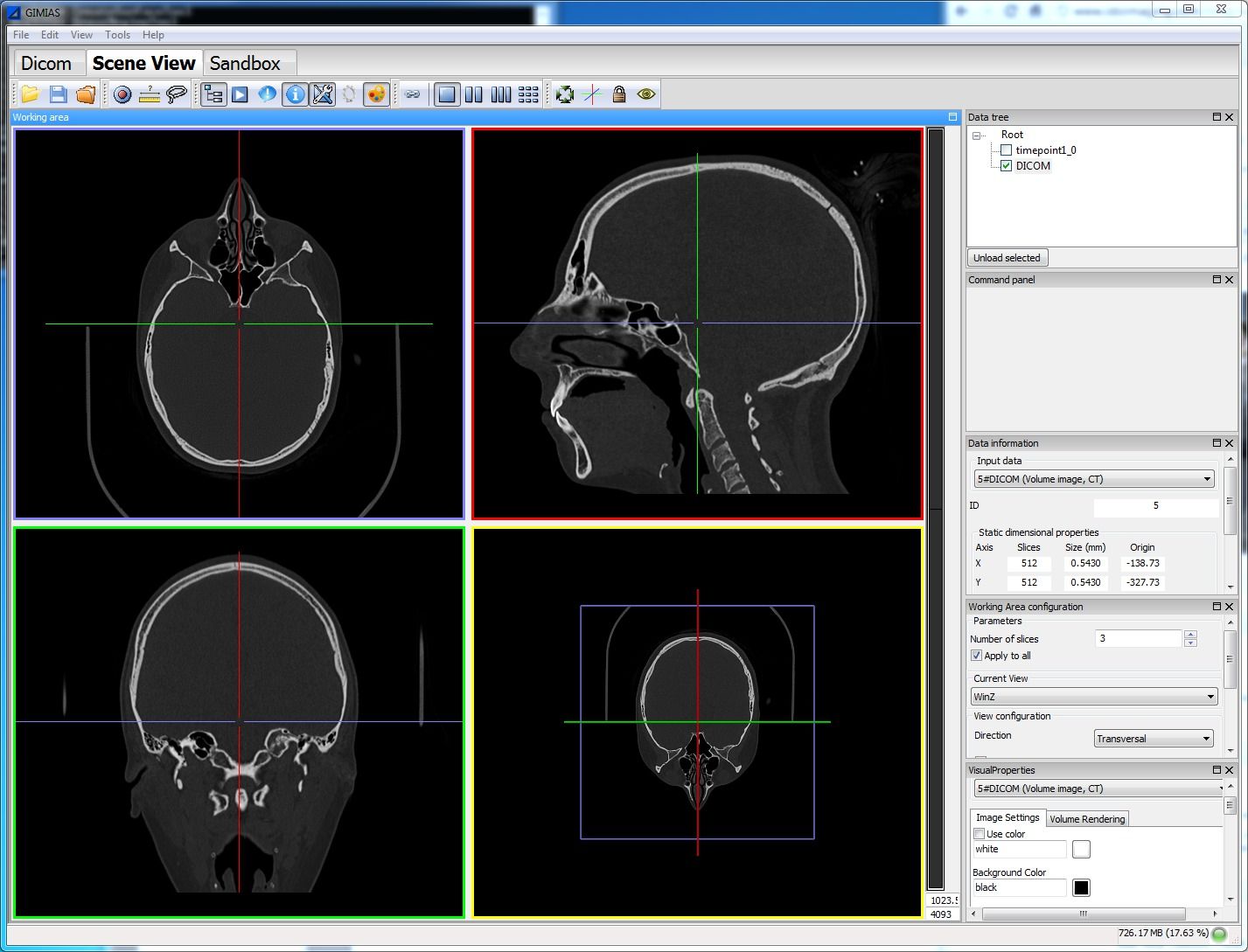
Gimias: is an open source framework for building medical Imaging & medical simulation applications for Windows and Linux
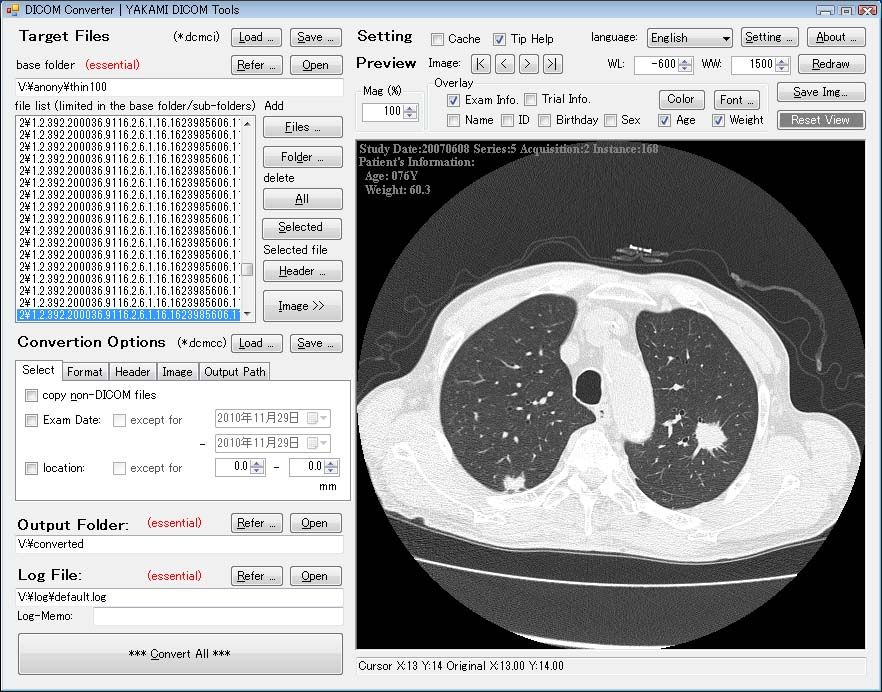 Yakami Converter
Yakami Converter
YAKAMI DICOM tools is a free lightweight DICOM viewer, & converter for Windows, we have reviewed it here.
Highlights
- Lightweight
- Easy to install, & use
- Works smoothly on old and new machines/ systems
- Suitable for novice users, & advanced users
- Supported languages: English, Japanese
20- Navegatium: (Windows)
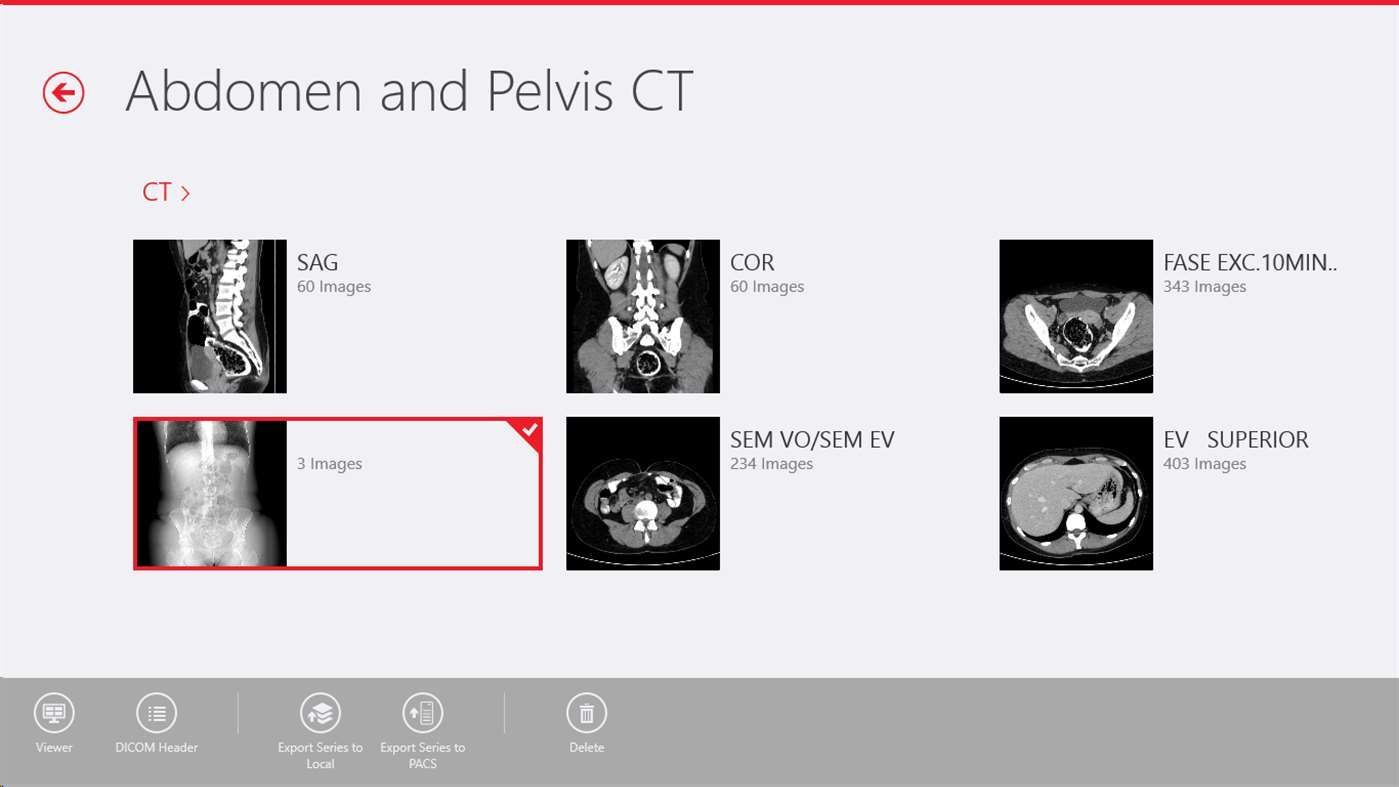 Navegatium
Navegatium
Navegatium is a Free 2D/3D DICOM Viewer with 3D Printing support for Windows. It’s full-featured DICOM viewer and PACS workstation for Windows, it supports Windows 7 and later.
Navegatium comes with full touch support for Windows-tablets, We have written a full review about it in here.
Highlights:
- DICOM / PACS Client
- 3D Printing on demand
- 3D Volume rendering
- Reports generation
- Navegatium Knowledge Base: Huge library of DICOM images
Navegatium’s Homepage
21- JimboDICOMViewer

JimboDICOMViewer is a free 2D/ 3D DICOM viewer to display and manage DICOM datasets and databases. It has unique features, but it’s available only for Linux and macOS.
Read more about JimboDICOMViewer
Hospital Information Systems (HIS)/ EMR with DICOM viewer
Some Hospital Information systems are built with DICOM viewer to ensure the usability for DICOM files. Here is a list of free open source hospital information systems some comes with complete DICOM and PACS integration.
Read also:
- 10+ Open source DICOM viewers and workstations ( Windows, Linux and Mac OSX )
- 20 Free & open source DICOM viewers for Windows
- 10+ Free Open source DICOM viewers for Mac OSX
- Horos Free/ Open source DICOM Viewer for Mac OSX for Radiologists.
- 10 Free iPhone and iPad DICOM & Radiology Applications for Doctors/ Radiologists
- 10 Open source DICOM/ PACS Servers
- Top 10 Open source & free HIS( Hospital Information systems) and EHR solutions
- Best 7 Free/ open source personal finance & accounting Manager apps for Windows, Mac OSX & Linux
- Best 20+ open source free accounting solutions for Windows, Linux, Mac OSX, and the cloud
- Sync.com — Like Dropbox, but secure, and 100% private.
Источник
Добрый день, хабрасообщество. Мне хотелось бы продолжить рассмотрение аспектов реализации DICOM Viewer’а, и сегодня речь пойдёт о функциональных возможностях.
Итак, поехали.
Инструментарий в 2D
Мультипланарная реконструкция (MPR)
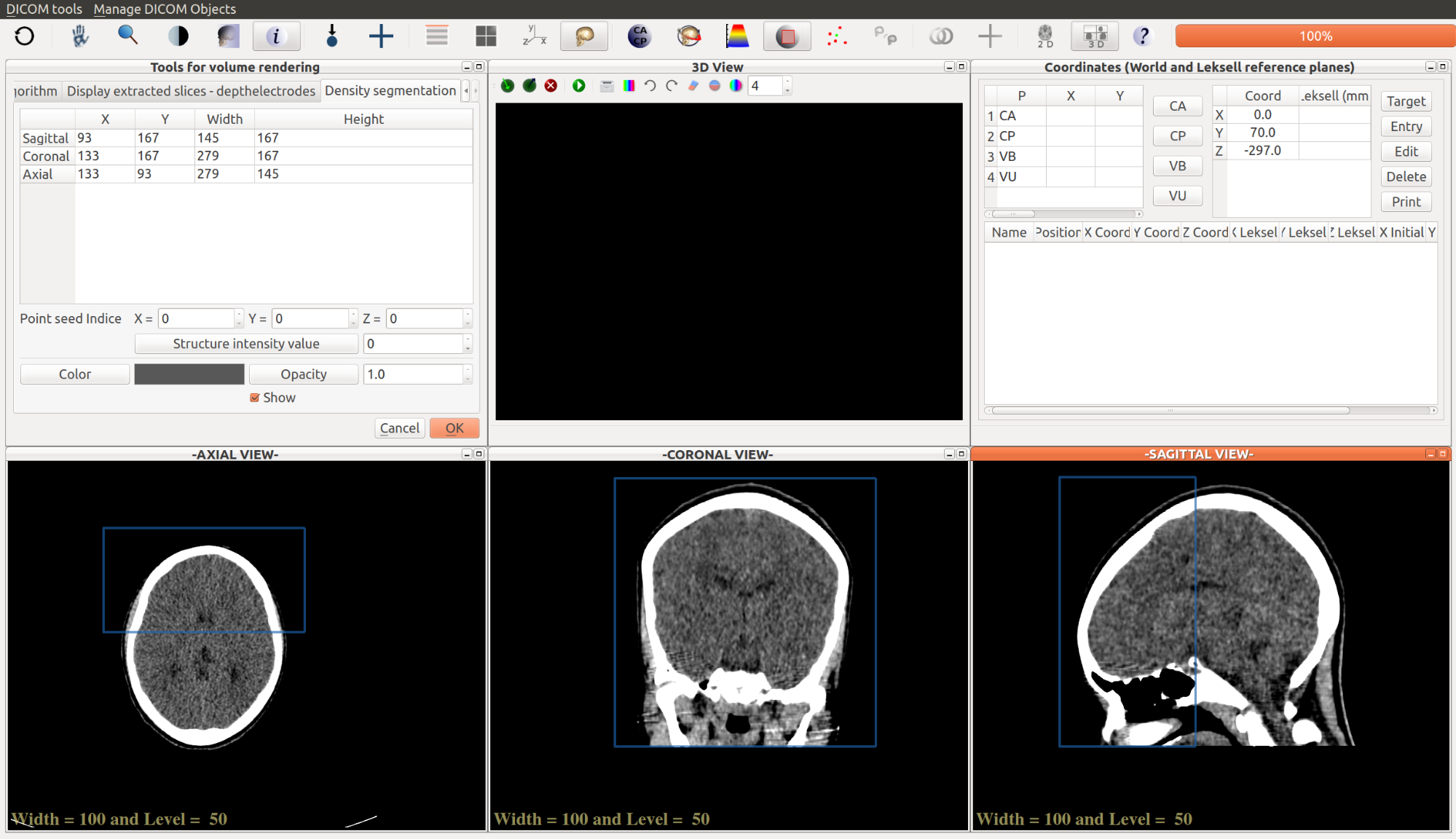
Мультипланарная реконструкция позволяет создавать изображения из оригинальной плоскости в аксиальную, фронтальную, сагиттальную или произвольную плоскости. Для того чтобы построить MPR, необходимо построить объёмную 3D-модель и «разрезать» её в нужных плоскостях. Как правило, наилучшее качество MPR получается при компьютерной томографии(КТ), потому что в случае КТ можно создать 3D модель с разрешением, одинаковым во всех плоскостях. Поэтому выходное MPR получается с таким же разрешением, какое было у исходных изображений, полученных из КТ. Хотя бывают и МРТ с хорошим разрешением. Вот пример мультипланарной реконструкции:
Зелёным — аксиальная плоскость (слева вверху);
Красным — фронтальная плоскость (справа вверху);
Синим — сагиттальная плоскость (слева внизу);
Жёлтым — произвольная плоскость (справа внизу).
Положение правого нижнего снимка определяется жёлтой линией на виде сбоку (левый верхний). Это и есть изображение, полученное «разрезанием» 3D-модели наклонной плоскостью. Для получения значения плотности в конкретной точки плоскости используется трилинейная интерполяция.
Мультипланарная реконструкция по произвольной кривой (curved MPR)
То же самое, что и MPR, только вместо произвольной плоскости можно взять кривую, как показано на рисунке. Используется, например, в стоматологии для панорамного снимка зубов.
Каждая точка на кривой задаёт исходную точку трассировки, а нормаль к кривой в этой точке соответствует направлению оси Y в двухмерном изображении для этой точки. Оси X изображения соответствует сама кривая. То есть в каждой точке двухмерного изображения направление оси X – это касательная к кривой в соответствующей точке на кривой.
Проекция минимальной/средней/максимальной интенсивности (MIP)
Значения минимальной интенсивности показывают мягкие ткани. Тогда как значения максимальной интенсивности соответствуют наиболее ярким участкам трёхмерного объекта — это либо наиболее плотные ткани, либо органы, насыщенные контрастным веществом. Минимальное/среднее/максимальное значение интенсивности берётся в диапазоне (как показано на рисунке пунктирными линиями). Минимальное значение по всей модели будет принимать воздух.
Алгоритм вычисления MIP очень простой: выбираем плоскость на 3D модели — пусть будет плоскость XY. Потом проходим по оси Z и выбираем максимальное значение интенсивности на заданном диапазоне и отображаем его на 2D плоскости:
Изображение, полученное путём проекции средней интенсивности, близко к обычному рентгеновскому снимку:
Некоторые виды радиологических исследований не дают должного эффекта без использования контрастного препарата, поскольку не отражают некоторые виды тканей и органов. Это связано с тем, что в организме человека есть ткани, плотность которых примерно одинакова. Чтобы отличать такие ткани друг от друга, используют контрастное вещество, которое придаёт крови большую интенсивность. Также контрастное вещество используется для визуализации сосудов при ангиографии.
Режим DSA для ангиографии
Ангиография — это приём, позволяющий визуализировать системы кровотоков (вены и сосуды) различных органов. Для этого используется контрастное вещество, которое вводят в исследуемый орган, и рентгеновский аппарат, создающий снимки во время ввода контрастного вещества. Таким образом на выходе аппарата получается набор снимков с разной степенью визуализации кровотоков:
Однако вместе с венами и сосудами на снимках видны ткани других органов, например, черепа. Режим DSA (Digital subtraction angiography) позволяет визуализировать только кровотоки без каких-либо других тканей. Как это работает? Берём изображение серии, в котором кровотоки ещё не визуализированы контрастным веществом. Как правило, это первое изображение серии, так называемая маска:
Затем вычитаем это изображение из всех остальных изображений серии. Получаем следующее изображение:
На этом изображении хорошо видны кровотоки и практически не видны другие ткани, что позволяет проводить более точную диагностику.
Инструментарий в 3D
Инструмент куб видимости (Clipping Box)
Инструмент Clipping Box позволяет увидеть кости и анатомические ткани в разрезе, а также показать внутренние органы изнутри. Инструмент реализуется на уровне рендера, просто ограничивая область рейтрейсинга.
В реализации область рейтрейсинга ограничивается плоскостями с нормалями, направленными в сторону отсечения. То есть куб представляется шестью плоскостями.
Инструментарий редактирования объема — вырезание многоугольником
Инструмент похож на предыдущий и позволяет удалять фрагмент объёма под произвольным многоугольником:
Под вырезанием следует понимать зануление вокселей в 3D-моделе, попавших в область многоугольника.
Также есть инструмент «Ножницы», который позволяют удалять части 3D-модели по принципу связности. Реализация: при выделении объекта происходит циклический поиск близлежащих связных вокселей, пока все близлежащие воксели не будут просмотрены. Затем все просмотренные воксели удаляются.
Линейка в 3D
В 3D можно производить измерения органов под любым углом, что невозможно для некоторых случаех в 2D.
В режиме 3D можно также воспользоваться полигональной линейкой:
Инструментарий в 4D
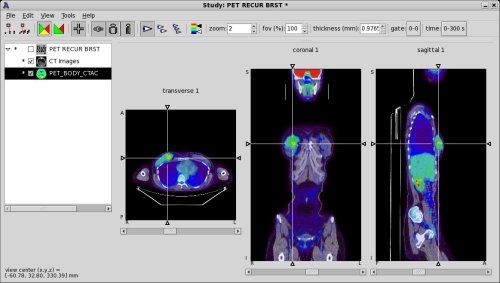
Совмещение нескольких томографических серий в 3D (Fusion PET-CT)
ПЭТ-КТ (англ. PET-CT) относительно новая технология, являющаяся исследовательским методом ядерной медицины. Является методом мультимодальной томографии. Четвёртым измерением в данном случае является модальность (PET и CT). Предназначена в основном для обнаружения раковых опухолей.
CT помогает получить анатомическую структуру человеческого тела:
а PET показывает определённые области концентрации радиоактивного вещества, которая напрямую связана с интенсивностью кровоснабжения данной области.
PET получает картину биохимической активности, детектируя в теле человека радиоактивные изотопы. Радиоактивное вещество скапливается в органах, насыщенных кровью. Затем радиоактивное вещество претерпевает позитронный бета-распад. Образовавшиеся позитроны в дальнейшем аннигилирует с электронами из окружающей ткани, в результате чего происходит излучение пар гамма-лучей, которые и детектируются аппаратом, и затем на основе полученной информации строится 3D изображение.
Выбор радиоактивного изотопа определяет биологический процесс, который желают отследить в процессе исследования. Процессом может быть метаболизм, транспорт веществ и др. Поведение процесса в свою очередь является ключом к верной диагностике заболевания. На изображении выше у пациента в области печени видна опухоль.
Но основываясь на PET трудно понять, в какой части тела находится область с максимальной концентрацией радиоактивного вещества. При соединении геометрии тела (CT) и областей, насыщенных кровью с высокой концентрацией радиоактивного вещества (PET), получаем:
В качестве радиоактивного вещества для PET применяются радиоактивные изотопы с разными периодами полураспада. Для образования всякого рода злокачественных образований используется фтор-18 (фтордезоксиглюкоза), йод-124 используется для диагностирования рака щитовидной железы, галлий-68 — для обнаружения нейроэндокринных опухолей.
Функционал Fusion формирует новую серию, в которой изображения обоих модальностей (и PET и CT) объединены. В реализации изображения обоих модальностей перемешиваются, а затем сортируются по оси Z (считаем, что X и Y – оси изображения). Фактически получается, что изображения в серии чередуются (PET, CT, PET, CT …). Эта серия в дальнейшем используется для отрисовки 2D fusion и 3D fusion. В случае 2D fusion изображения отрисовываются попарно(PET-CT) в порядке возрастания Z:
В данном случае сначала был отрисовано изображение CT, затем PET.
3D fusion реализован для видеокарты на CUDA. На видеокарте отрисовываются одновременно обе 3D-модели — PET и CT и получается реальный мультимодальный fusion. На процессоре fusion тоже работает, но работает несколько иначе. Дело в том, что на процессоре обе модели представлены в памяти как отдельные окто-деревья. Следовательно, при отрисовке необходимо трассировать два дерева и синхронизировать пропуск прозрачных вокселей. А это бы значительно снизило скорость работы. Поэтому было решено просто накладывать результат рендера одной 3D-модели поверх другой.
4D CardiacCT
Технология Cardiac CT используется для диагностики различных нарушений работы сердца, включая коронарную болезнь сердца, тромбоэмболия легочной артерии и другие заболевания.
4D Cardiac CT представляет собой 3D во времени. Т.е. получается небольшое видео, которое будем называть кинопетлёй, в которой каждый кадр будет представлять собой 3D-объект. Исходные данные представляют собой набор dicom-изображений сразу для всех кадров кинопетли. Для того чтобы преобразовать набор изображений в кинопетлю, необходимо сначала сгруппировать исходные изображения по кадрам, а затем для каждого кадра создать 3D. Построение 3D-объекта на уровне кадра происходит так же как и для любой серии dicom-изображений. Мы используем эвристическую сортировку изображений для группировки по кадрам, используя положение изображения на оси Z (считая что X и Y это оси изображения). Полагаем, что после группировки по кадрам, в каждом кадре получается одинаковое количество изображений. Переключение кадра фактически сводится к переключение 3D-модели.
5D Fusion Pet – CardiacCT
5D Fusion Pet – CardiacCT — это 4D Cardiac CT с добавлением fusion с PET в качестве пятой размерности. В реализации сначала создаём две кинопетли: с CardiacCT и с PET. Затем делаем fuision соответствующих кадров кинопетель, что даёт нам отдельную серию. Затем строим 3D полученной серии. Выглядит это так:
Виртуальная эндоскопия
В качестве примера виртуальной эндоскопии будем рассматривать виртуальную колоноскопию, поскольку она является наиболее распространённым видом виртуальной эндоскопии. Виртуальная колоноскопия позволяет на основе данных КТ построить объёмную реконструкцию области брюшной полости и по этой трёхмерной реконструкции произвести диагностику. Во вьюере есть инструмент полёт камеры (fly-through) с навигацией по MPR:
который в том числе позволяет автоматически следовать анатомической структуре. В частности позволяет просматривать внутрикишечную область в автоматическом режиме. Вот как это выглядит:
Полёт камеры представляет серию последовательных перемещений по внутрикишечной области. Для каждого шага вычисляется вектор перемещения камеры в следующую часть анатомической структуры. Вычисление производится на основе прозрачных вокселей в следующей части анатомической структуры. Фактически вычисляется некий средний воксель среди прозрачных. Начальный вектор перемещения задаётся вектором камеры. В инструменте Полёт камеры используется исключительно перспективная проекция.
Также есть функционал для автоматической сегментации кишечника, т.е. функционал для отделения кишечной области от остальной анатомии:
Возможна также навигация по сегментированной 3D-модели (кнопка Show camera orientation), которая по клику мыши на 3D-моделе перемещает камеру на соответствующую позицию в исходной анатомии.
Сегментация реализуется с помощью волнового алгоритма. Полагается, что анатомия замкнутая в том смысле, что она не контактирует с другими органами и внешним пространством.
Система просмотра ЭКГ (Waveform)
Отдельным модулем во viewer’е реализовано чтение данных из Waveform и их отрисовка. DICOM ECG Waveform это специальный формат хранение данных отведений электрокардиограмм, определяемый стандартом DICOM. Данные электрокардиограммы представляют собой двенадцать отведений — 3 стандартных, 3 усиленных и 6 грудных. Данные каждого отведения представляют собой последовательность измерений электрического напряжения на поверхности тела. Для того чтобы отрисовать напряжения, нужно знать масштаб по вертикали в мм/мВ и масштаб по горизонтали в мм/сек:
В качестве вспомогательных атрибутов также отрисовывается сетка для простоты измерения расстояний и масштаб в левом верхнем углу. Варианты масштаба подобраны с учётом врачебной практики: по вертикали — 10 и 20 мм/мВ, по горизонтали — 25 и 50 мм/сек. Также реализованы инструменты для измерения расстояния по горизонтали и вертикали.
DICOM-Viewer как DICOM-клиент
DICOM-Viewer, помимо прочего, представляет собой полноценный DICOM-клиент. Есть возможность производить поиск на PACS-сервере, получать из него данные и др. Функции DICOM-клиента реализованы с помощью открытой библиотеки DCMTK. Рассмотрим типичный use-case работы DICOM-клиента на примере viewer’а. Производим поиск стадий на удалённом PACS-сервере:
При выборе стадии внизу отображаются серии для выбранной стадии и количество изображений в них. Сверху справа указывается PACS-сервер, на котором будет произведён поиск. Поиск можно параметризовать, уточняя критерии поиска: PID, дата исследования, имя пациента и др. Поиск на клиенте реализуется командой C-FIND SCU с помощью библиотеки DCMTK, которая работает на одном из уровней: STUDY, SERIES и IMAGE.
Далее изображения выбранной серии можно загрузить, используя команды С-GET-SCU и C-MOVE-SCU. Протокол DICOM обязывает стороны соединения, т.е. клиента и сервера, заранее договориться, какие типы данных они собираются передавать через это соединение. Под типом данных понимается комбинация значений параметров SOPClassUID и TransferSyntax. SOPClassUID определяет тип операции, которую планируется выполнять через данное соединение. Наиболее часто используемые SOPClassUID’ы: Verification SOP Class (пинг сервера), Storage Service Class (сохранение изображений), Printer Sop Class (выполнение печати на DICOM-принтере), CT Image Storage (сохранение изображений КТ), MR Image Storage (сохранение изображение МРТ) и другие. TransferSyntax определяет формат бинарного файла. Популярные TransferSyntax’ы: Little Endian Explicit, Big Endian Implicit, JPEG Lossless Nonhierarchical (Processes 14). То есть, чтобы передать МРТ изображения в формате Little Endian Implicit, то в соединение необходимо добавить пару MR Image Storage — Little Endian Explicit.
Загруженные изображения сохраняются в локальное хранилище и, при повторном просмотре, загружаются из него, что позволяет увеличить производительность viewer’а. Сохранённые серии помечаются жёлтым значком в верхнем левом углу первого изображения серии.
Также DicomViewer как DICOM-клиент умеет записывать диски с исследованиями в формате DICOMDIR. Формат DICOMDIR реализуется в виде бинарного файла, который содержит относительные пути ко всем DICOM-файлам, которые записываются на диск. Реализуется с помощью библиотеки DCMTK. При чтении диска считываются пути ко всем файлам из DICOMDIR и после этого загружаются. Для добавления в DICOMDIR стадий и серий был разработан такой интерфейс:
Вот и всё, что я хотел рассказать про функционал DicomViewer’а. Как всегда очень приветствуется обратная связь от квалифицированных специалистов.
Ссылка на Viewer:
DICOM Viewer x86
DICOM Viewer x64
Примеры данных:
MANIX — для общих примеров (MPR, 2D, 3D и т.д.)
COLONIX — для виртуальной колоноскопии
FIVIX — 4D CARDIAC-CT
CEREBRIX — Fusion PET-CT
Источник
